Vorstellung des Projekts
Das DeepAnatomy Projekt beschäftigt sich mit Machine Learning Algorithmen, insbesondere Deep Learning, zur Analyse und Auswertung medizinischer Bilder. Dabei handelt es sich um ein zwei-semestriges Studentenprojekt, das durch eine Kooperation der Universität Bremen und dem Fraunhofer-Institut für Digitale Medizin MEVIS ermöglicht wird. Im Wintersemester 2019 wurde das Projekt erstmalig für Bachelorstudierende angeboten, im Wintersemester 2020 folgte das darauf aufbauende Masterprojekt.
Deep Learning in der medizinischen Bildgebung
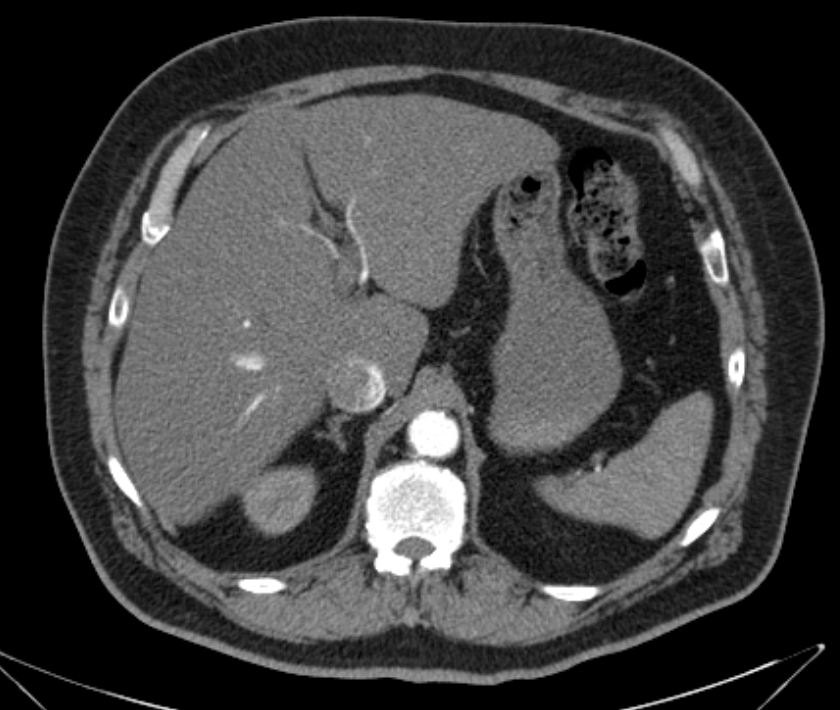
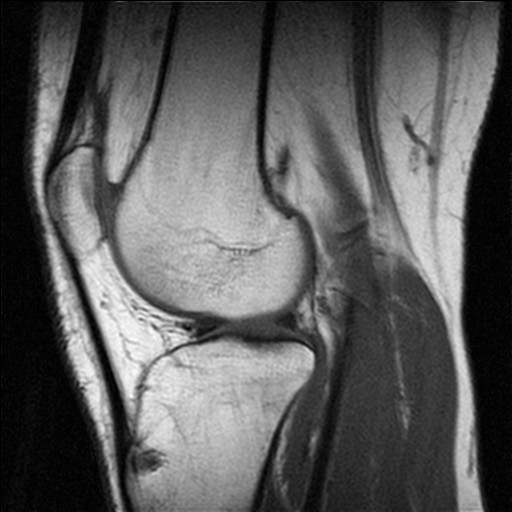
Die Teilnehmer des Projekts setzen sich mit den aktuellen, innovativen Deep Learning Algorithmen auseinander und wenden diese auf medizinischen Bildern an. Dazu zählen vor allem Bilder der Computertomographie (CT), aber auch der Magnetresonanztomographie (MRT), Ultraschall, Röntgen und weitere Methoden der medizinischen Bildgebung.
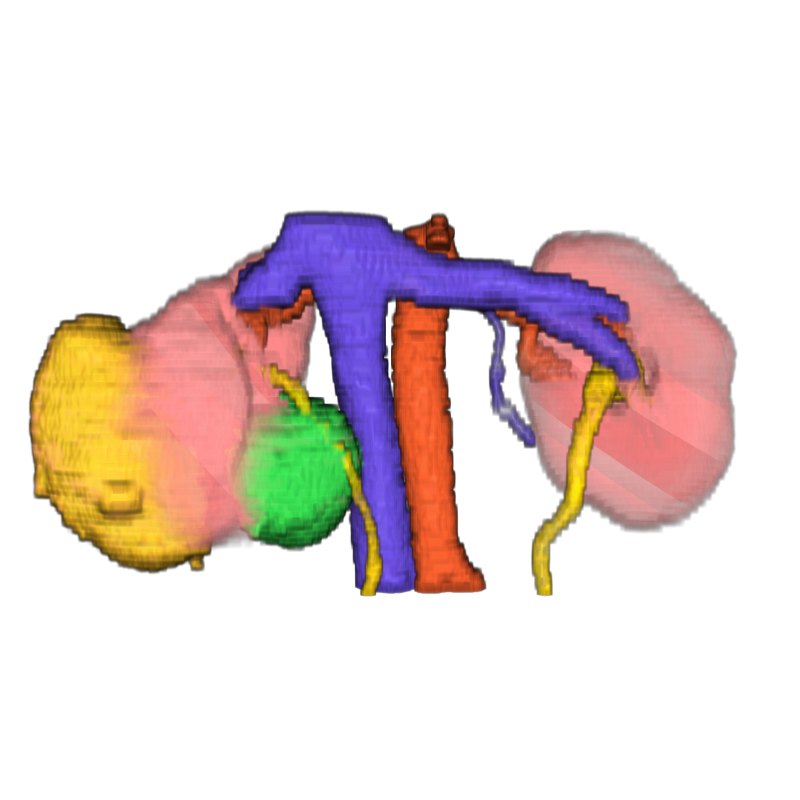
Mit Hilfe von Deep Neural Networks werden die medizinischen Bilder hinsichtlich eines festgelegten Ziels ausgewertet. Häufig sollen bestimmte Bildbereiche, wie anatomische Strukturen, Anomalien usw., segmentiert werden, um so beispielsweise eine Diagnose zu ermöglichen bzw. zu erleichtern. Darüber hinaus gibt es jedoch eine Vielzahl von Einsatzmöglichkeiten von Deep Learning in der medizinischen Bildgebung, wie das Verbessern der Bildqualität oder die Synthese und Augmentation von Bildern.
Da es sich bei medizinischen Bildern um eine Domäne handelt, die sich stark von herkömmlichen Bildern, z.B. einer Handykamera, abhebt, haben sich dafür spezielle Deep Learning Methodiken entwickelt. Diese beinhalten unter anderem ein besonderes Pre-/Postprocessing der Bilder sowie eigene Architekturen der Neuronalen Netze, z.B. U-Net und DeepMedic, die sich insbesondere für medizinische Bilder eignen.
Infrastruktur
MEVIS bietet Soft- und Hardwareressourcen an, die die Arbeit mit Deep Learning deutlich verbessern. Zum Trainieren steht ein Rechnercluster und Netzwerkspeicher zur Verfügung, sodass auch mit einfachen PCs gearbeitet werden kann.
In einer umfangreichen Python Bibliothek stehen die gängigsten Deep Learning Archtikturen und Komponenten der medizinischen Bildgebung, wie bspw. U-Nets, zur Verfügung und ermöglichen eine einfache Anwendung. Darin werden die aktuell größten Frameworks Tensorflow/Keras/Pytorch zur Implementierung verwendet.
Zusätzlich gibt es ein dutzend Tools, die die Arbeit mit medizinischen Daten erleichtern. Für Pre-/ Postprocessing und Visualisieren von Bilddaten ist die graphische Programmierumgebung MeVisLab sehr geeignet und bietet Schnittstellen zum Training und Inferenz der Architekturen. Intern bauen hier C++ und Python die graphischen Module auf und kommunizieren per RPC Verbindung mit Tensorflow/Pytorch Instanzen.
Zudem steht die Webapp Challengr zum Vergleich der Ergebnisse der verschiedenen Neuronalen Netzen zur Verfügung. Diese bietet eine übersichtliche Darstellung und Diagramme der Trainingsparameter sowie der Evaluationsergebnisse.
Zum besseren Verständnis, welche Möglichkeiten und Tools es gibt, ein Beispiel aus dem aktuellen Projekt: Wir haben neue Architekturen in Keras implementiert und in MeVisLab eingebunden. Um diese auf dem Cluster zu nutzen, mussten wir eigene Docker Images bauen und bei einem MEVIS Registry Service hochladen. Über ein Vue.js/Quasar Dashboard konnten wir dann ein Training starten. Über dieses Cluster läuft auch Challengr, welches all unsere trainierten Netze und dazugehörigen Einstellungen auflistet und auswertet. Durch Docker Container und Quasar Webapps kann sehr viel über das Netzwerk gemacht werden, was auch in der Remote Arbeit hilfreich ist.
Die Projekt-Organisation und Dokumentation findet in Jira, GitLab und Confluence statt.
Aufgabenstellung und Ziele
Die bisherigen DeepAnatomy Projekte orientierten sich an einer öffentlichen Deep Learning Challenge. Anhand dieser konnten folgende Themen und Aufgaben behandelt werden:
- Kennenlernen der allgemeinen Deep Learning Methodiken: Pre-/Postprocessing, Training, Inferenz/Evaluation
- Kennenlernen der medizinspezifischen Deep Learning Methodiken: spezielles Pre-/Postprocessing in MeVisLab, besondere Deep Learning Architekturen und Konfigurationen, usw.
- Gemeinsames Anwenden und Vergleichen verschiedener Neuronaler Netze im Rahmen einer öffentlichen Challenge (z.B. KiTS Challenge: kits19 grand-challenge)
- Erweitern der vorhandenen MEVIS Infrastruktur um beispielsweise weitere Netzwerk-Architekturen, Evaluationsmethodiken usw.
Grundsätzlich haben die Teilnehmer große Freiheiten in der Auswahl ihrer Tätigkeiten und können sich so auf ihre Interessen fokussieren. Natürlich sollte ein wissenschaftlicher Zusammenhang zu den allgemeinen Zielen des Projekts bestehen.
Vorkentnisse und Anforderungen
Es werden keine besonderen Vorkenntnisse der Teilnehmer erwartet. Da sich das Projekt auf das Thema Deep Learning fokussiert, sind Kenntnisse in diesem Bereich hilfreich und erleichtern den Projekteinstieg. Diese sind jedoch nicht zwingend erforderlich.
Zusammenfassend sind folgende Vorkenntnisse hilfreich (aber nicht erforderlich):
- Python, inkl. Tensorflow, Keras und PyTorch
- MeVisLab
- Docker Container
- Vue.js
